Image credit: CDC Unsplash
Estimando R_0 de Granada (ritmo de contagio) en ventanas semanales
En lugar de hacer estimaciones o predicciones (aún creo que tengo que seguir leyendo más sobre el tema para lanzarme) he optado por analizar el ritmo reproductivo básico.
(de Wikipedia) En epidemiología, el número básico de reproducción (a veces llamado ritmo básico de reproducción, ratio reproductiva básica y denotadas por $R0$, r sub-cero) de una infección es el número promedio de casos nuevos que genera un caso dado a lo largo de un período infeccioso.
Este número es importante porque permite darnos una idea de cómo está evolucionando el virus y si está siendo útil las normas de confinamiento. Además nos ayuda a evaluar la evolución de la pandemia: cuando el numéro baje de 1 tendremos un virus que se contagia a menos de una persona, es decir que tenderá a desaparecer en el tiempo.
Así pues, vamos a analizar el $R_0$ en la provincia en la que resido durante esta epidemia: Granada.
Getting the data
A la hora de obtener los datos, he ido directamente a la página de la Junta de Andalucía. Es cierto que la página está un pelín atrasada, pero es la única fuente oficial que he encontrado que permite personalizar los datos para obtener los de Granada en particular.
La descargar de datos es bastante sencilla, y al poco tengo un csv con los casos nuevos, los ingresos en UCI y las muertes.
En nuestro caso, vamos a usar los casos nuevos. Evidentemente tenemos sesgos debido a que nuestro país no esta realizando test masivos, aún así, para evaluar la pandemia son interesantes.
También tenemos que tener en cuenta que Granada es una ciudad típicamente universitaria, y que a poco de comenzar el confinamiento, muchos universitarios han regresado a su casa. Esto creo que también ayuda a la evolución de la enfermedad en esta provincia.
Analyzing the data
Para anailzar los datos vamos a cambiar de Python ( mi lenguaje habitual) a R. ¿Por qué? Bueno esencialmente por comodidad. Realmente R es un lenguaje bastante sencillo de manejar y posee un gran cantidad de paquetes que hacen muy sencillo el análisis estadístico de datos de variados orígenes.
En nuestro caso vamos a usar EpiEstim, paquete que por ejemplo, también está usando ElPais (un periodico español) en sus análisis. EpiEstim se encuentra en los repositorios oficiales de R y podéis encontrar el artículo que lo presenta aqui: A New Framework and Software to Estimate Time-Varying Reproduction Numbers During Epidemics Anne Cori, Neil M. Ferguson, Christophe Fraser and Simon Cauchemez American Journal of Epidemiology 2013.
Este paquete se centra en el estudio de $R_t$, esat constante es el número reproductivo instantaneo, que se puede estimar mediante el número de nuevas infecciones generadas en tiempo $t$, frente al total de infecciosidad de los individuos contagiados en tiempo $t$. Este total se calcula siguiendo un sumatorio que usa la distribución de probabilidad que sigue la enfermedad ( que suponemos normal) y la incidencia en $t-1$. En resumen, $R_t$ es la media de casos secundarios que cada individuo contagiado puede provocar si las condiciones se mantienen en tiempo $t$.
Hacer inferencia sobre esta variable puede ser complicado por su alta variabilidad. Por eso, el paquete plantea hacer inferencia de la misma en ventanas temporales que se solapan, para aportar mayor significación a las estimaciones.
Lo último que necesitamos para hacer nuestra predicción es el intervalo serial (serial interval en ingles). El intervalo serial es el nombre que recibe el tiempo que transcurre entre los casos sucesivos en una cadena de transmisión.
Habitualmente se estima generalmente a partir del intervalo entre los inicios clínicos, en cuyo caso es el “intervalo de serie de inicio clínico” cuando estas cantidades son observables. En principio, podría estimarse por el intervalo de tiempo entre la infección y la transmisión posterior. En el caso particular del covid he usado esta referencia:
- Serial interval of novel coronavirus (COVID-19) infections. Hiroshi Nishiura, Natalie M. Linton,Andrei R. Akhmetzhanov
Con lo que el intervalo será de 4.6 días con media de 4.8 días y desviación típica de 2.3 días.
Plot
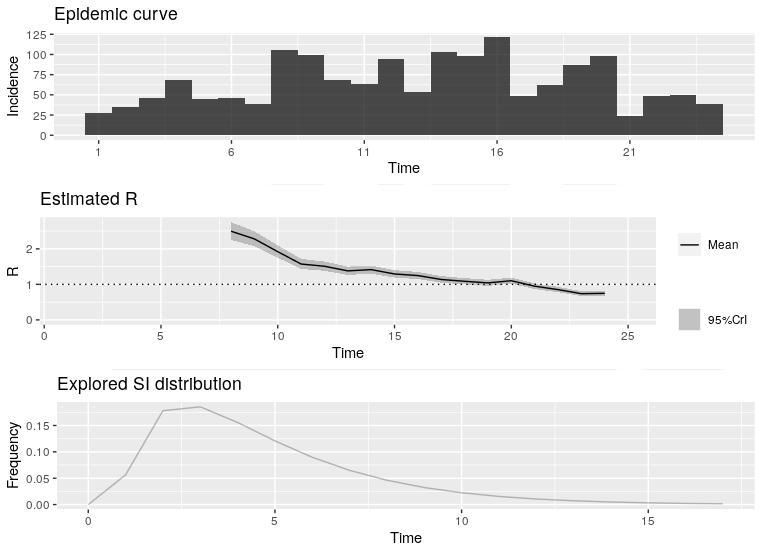
Obtenemos finalmente el gráfico. Como véis he representado 3 datos esenciales para nuestro análisis. Por una parte un histograma básico con el número de casos que han acontecido estos días (desde que fueron representativos, recordemos que Granada tuvo 0 casos hasta muy entrada la pandemia en muchas provincias).
En segundo lugar aparece la estimación del ritmo reproductivo básico a lo largo del tiempo. Aunque no se aprecie posee los rangos superior e inferior del mismo. Y finalmente se muestra cual es el intervalo serial escogido como hemos comentado en la sección anterior.
Conclusions!
Parecen buenas noticias! Hemos pasado ya a un estado con $R_0<1$, aunque son buenas noticias (implicarían que a la larga la enfermedad desaparecería) hay que tener en cuenta que esto se debe a las medidas de confinamiento y que debemos ser muy cautos aún.
Deberíamos bajar este término todo lo posible antes de acabar con el confinamiento, para reducir lo máximo posible los rebrotes.
A