COVID19 Kaggle Challenge
Dentro de los challenges lanzados por Kaggle para usar los datos publicados del COVID19, los doctorandos que compartimos directora de tesis nos juntamos para probar ideas y aprender lo que pudiésemos en el camino.
Había varios retos o tareas en las que podíamos embarcarnos, pero como nuestro interés era más bien académico, decidimos empezar haciendo un análisis exploratorio de datos y clusterización. Y desde ahí, lo que quisiésemos o nos diese tiempo.
Los resultados que véis aquí corresponden a un enfoque muy sencillo y básico de aplicación de técnicas de machine learning para crear un recomendador de artículos científicos relacionados con uno seleccionado por el usuario.
El objetivo es usar los titulos y abstracts de los artículos para extraer “de qué van”. Una vez obtenido esto, se buscan artículos que traten temas similares.
Para ello seguimos estos pasos:
- EDA
- Text Preprocessing
- Tokenization
- vectorization
- LDA and Topic Extraction
- Recommendation system
Si quereis echarle un vistazo a el notebook (aunque esta desordenado): https://www.kaggle.com/thebooort/covid19-exploration-and-paper-recommendation
Este post pretende presentar los principales resultados del notebook (hasta que encuentre una forma de subir los notebooks enteros), omitiré trozos de código que considere poco importantes.
EDA
En este aspecto, trabajando con abstract y texto, tendremos que saber cuantas palabras realmente van a entrar a nuestro sistema. Para ello una diagramas de cajas puede sernos útil:
|
|
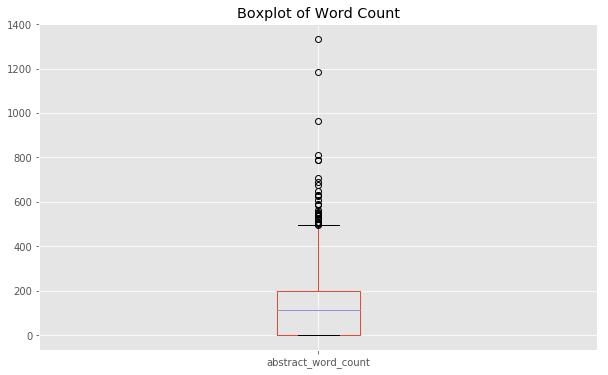
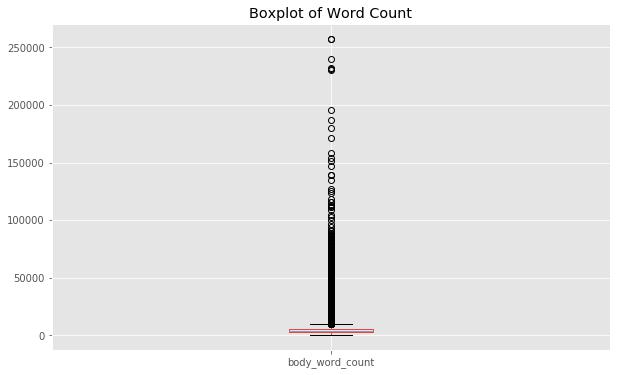
Además, una nube de palabras tambien puede darnos una idea aproximada de qué palabras tienen importancia (tanto aquellas que son importante como las que no).
|
|



Tokenización
Para esta seccion siguiendo un código de ajrwhite, usamos scapy. La idea es tokenizar el texto, no solo para esta parte, si no para cualquier analisis o algoritmo posterior, quedandonos con un texto depurado y palabras que realmente sean importante para etiquetar, clasificar, o mdelizar estos textos.
|
|
Del word cloud tenemos algunas sugerencias de stop words que no son relevantes en nuestro análisis.
Añadiéndolas lanzamos finalmente la tokenizacion y la aplicamos sobre nuestras columnas de texto.
|
|
Vectorización
Vamos a vectorizar nuestra información textual ( esto es, transformar las palabras en vectores numéricos). Para ello podemos usar CountVectorizer o TF-IDF entre otros. Usé estos dos por simpleza, pero CountVectorizer me dió mejores resultados (CountVectorizer esencialmente vectoriza la palabra contando la frecuencia de repetición de esa palabra en los títulos o en los abstracts).
Importando sklearn usar cualquiera de estos dos algoritmos es muy sencillo. Además, podemos añadir el tokenizador que queramos, en este caso el que hemos construido con spacy.
|
|
LDA
LDA es basciamente un método estadístico que busca encontrar una combinación lineal de rasgos que caracterizan o separan dos o más clases de objetos o eventos. La combinación resultante la podemos utilizar para separar nuestros textos en grupos y obtener qué palabras aparecen con más frecuencia dentro de esos grupos, a fin de caracterizarlos.
|
|
Resultado final del LDA
Para visualizar LDA a parte de obtener los topics, usé pyLDAvis. pyLDAvis es una biblioteca de Python para visualización interactiva de topic models (basada en el paquete R de Carson Sievert y Kenny Shirley).
Como dicen en su web pyLDAvis está diseñado para ayudar a los usuarios a interpretar los topics en un modelo de topics que se ha ajustado a un corpus de datos textuales. El paquete extrae la información de un modelo de topics LDA ajustado, para ofrecer una visualización interactiva.
Os lo recomiendo.
|
|
La visualización está diseñada para ser utilizada en cualquier notebook, pero también se puede guardar en un archivo HTML independiente para compartir fácilmente:
|
|
Podéis consultar el resultado como una página aparte aqui: https://thebooort.github.io/covizzz19/ , así os quitáis de problemas si estáis viendo este blog como theme oscuro.
Visualizando las temáticas
Las temáticas finales obtenidas fueron:
Topic 0 ['protein', 'rna', 'proteins', 'activity', 'cell', 'replication', 'antiviral']
Topic 1 ['infection', 'cell', 'immune', 'infected', 'mice', 'response', 'induced', 'expression']
Topic 2 ['respiratory', 'pcr', 'samples', 'detection', 'viruses', 'assay', 'using', 'positive']
Topic 3 ['diseases', 'disease', 'review', 'development', 'health', 'research', 'based', 'new', 'use', 'infectious']
Topic 4 ['patients', 'calves', 'il', 'group', 'associated', 'days', 'cats', 'clinical', 'age']
Topic 5 ['cov', 'sars', 'protein', 'coronavirus', 'sequence', 'mers', 'human', 'genome', 'viruses']
Topic 6 ['health', 'influenza', 'risk', 'data', 'cases', 'disease', 'outbreak']
Podemos estudiar las correlaciones entre las puntuaciones de los topics de cada paper:
|
|
Obtenemos una correlación baja entre topics, lo que implica que nuestra clusterizacion (si bien mejorable) no es mala.
Recomendaciones
Para obtener la recomendacion final, bastará con comparar las componentes que identifican a cada paper. Seleccionado uno cualquiera, calcularemos la distancia con el resto de papers y devolveremos los mas cercanos. Esta distancia la podemos definir como queramos: RMSE, Cosine similarity, etc.
|
|
Probamos un caso concreto
|
|